使用参数化 UMAP 转换新数据
在许多情况下,您可能希望使用现有的 UMAP 模型,并将新数据嵌入到学到的空间中。对于一个简单的例子,其中高维训练数据的整体分布与新嵌入数据的分布相匹配,请参阅 使用 UMAP 转换新数据。然而,我们不能总是确定情况会是这样。为了模拟我们想要在嵌入空间中包含新颖行为的情况,我们将使用 MNIST 数字数据集(参见 如何使用 UMAP 获取基本示例)。
要跟随此示例,请参阅 GitHub 仓库中的 MNIST_Landmarks notebook。
import keras
from sklearn.model_selection import train_test_split
from umap import UMAP, ParametricUMAP
import matplotlib.pyplot as plt
import numpy as np
import pandas as pd
我们将首先加载数据集,并使用 sklearn 的 train_test_split 函数将其分成两个相等的部分。这将为我们提供两个分区进行处理,一个用于训练我们的原始嵌入,另一个用于测试。为了模拟数据中出现的新行为,我们从 x1 分区中删除一个 MNIST 类别 N。在这种情况下,我们将使用 N=2,因此我们的模型将在除 2 以外的所有数字上进行训练。
(X, y), (_, _) = keras.datasets.mnist.load_data()
x1, x2, y1, y2 = train_test_split(X, y, test_size=0.5, random_state=42)
# Reshape to 1D vectors
x1 = x1.reshape((x1.shape[0], 28*28))
x2 = x2.reshape((x2.shape[0], 28*28))
# Remove one category from the train dataset.
# In the case of MNIST digits, this will be the digit we are removing.
N = 2
x1 = x1[y1 != N]
y1 = y1[y1 != N]
print(x1.shape, x2.shape)
(26995, 784) (30000, 784)
使用 UMAP 处理新数据
首先,我们将确定在这种情况下直接使用 UMAP 的问题,然后我们将看到如何使用参数化 UMAP 来解决这些问题。首先,我们需要在 x1 分区上训练一个 UMAP 模型。
embedder = UMAP()
emb_x1 = embedder.fit_transform(x1)
可视化我们的结果
plt.scatter(emb_x1[:,0], emb_x1[:,1], c=y1, cmap='Spectral', s=2, alpha=0.2)
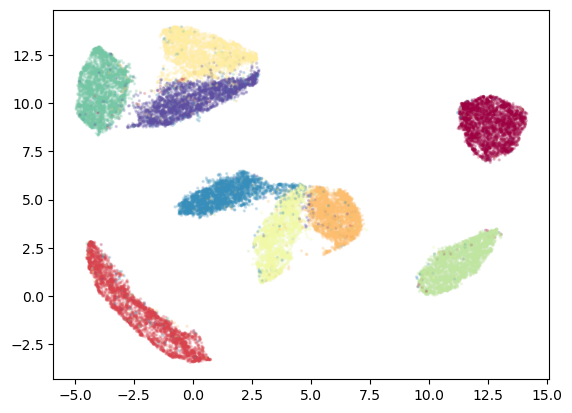
这是一个干净且成功的嵌入,正如我们在这个相对简单的示例中对 UMAP 的预期一样。我们看到了嵌入 MNIST 时通常会看到的结构,但没有任何数字 2。 UMAP 类构建为与 scikit-learn 兼容,因此通过传递新数据就像使用 transform 方法并传递新数据一样简单。我们将通过 x2,其中包含原始类别的未见示例,以及我们保留类别 N(数字 2)的样本。
为了让类别 N 的样本更显眼,我们将它们用黑色叠加绘制。
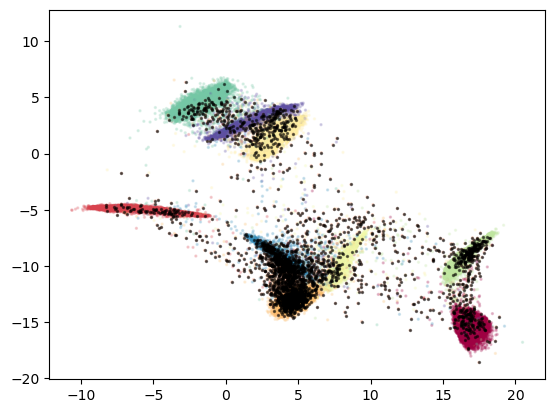
emb_x2 = embedder.transform(x2)
plt.scatter(emb_x2[:,0], emb_x2[:,1], c=y2, cmap='Spectral', s=2, alpha=0.2)
plt.scatter(emb_x2[y2==N][:,0], emb_x2[y2==N][:,1], c='k', s=2, alpha=0.5)

尽管我们的 UMAP 嵌入器正确处理了 x1 中存在的类别,但它对我们保留类别 N 的示例处理得不好。这些点中的许多集中在现有类别之上,有些则分散在它们之间。这种泛化能力不足并非 UMAP 独有,而是 learned embeddings 的一个普遍难题。这也可能或可能不是一个问题,具体取决于您的使用场景。
使用参数化 UMAP 处理新数据
我们可以使用参数化 UMAP 改善这一结果。参数化 UMAP 与 UMAP 的不同之处在于,它使用神经网络学习数据与嵌入之间的关系,而不是直接学习嵌入。这意味着我们可以通过继续训练神经网络,更新权重来整合新信息,从而包含新数据。
有关参数化 UMAP 及其提供的众多选项的更完整信息,请参阅 参数化(神经网络)嵌入。
我们将通过训练一个 ParametricUMAP 嵌入模型并运行相同的实验来开始解决这个问题。
p_embedder = ParametricUMAP()
p_emb_x1 = p_embedder.fit_transform(x1)
plt.scatter(p_emb_x1[:,0], p_emb_x1[:,1], c=y1, cmap='Spectral', s=2, alpha=0.2)

同样,我们在对 x1 的初始嵌入上获得了良好的结果。如果我们不重新训练就通过 x2,我们会遇到与我们的 UMAP 模型类似的问题。
p_emb_x2 = p_embedder.transform(x2)
plt.scatter(p_emb_x2[:,0], p_emb_x2[:,1], c=y2, cmap='Spectral', s=2, alpha=0.2)
plt.scatter(p_emb_x2[y2==N][:,0], p_emb_x2[y2==N][:,1], c='k', s=2, alpha=0.5)
使用地标重新训练参数化 UMAP
为了更新我们的嵌入以包含新类别,我们将对现有的 ParametricUMAP 模型进行微调。如果不进行其他任何更改,这将从我们上次停止的地方开始,但我们的嵌入空间的结构可能会漂移和改变。这是因为 UMAP 损失函数对平移和旋转是不变的,它只关注点之间的相对位置和距离。
为了保持我们的嵌入空间更加一致,我们将使用 ParametricUMAP 的 landmarks 选项。我们在 x2 分区上重新训练模型,同时包含从 x1 中选择的一些点作为地标。我们将选择 x1 中 1% 的样本包含进来,并使用它们在嵌入空间中的当前位置作为 landmarks 损失函数的一部分。
默认的 landmark_loss_fn 是点原始位置和当前位置之间的欧几里得距离。我们将做的唯一改变是设置 landmark_loss_weight=0.01。
# Select landmarks indexes from x1.
#
landmark_idx = list(np.random.choice(range(x1.shape[0]), int(x1.shape[0]/100), replace=False))
# Add the landmark points to x2 for training.
#
x2_lmk = np.concatenate((x2, x1[landmark_idx]))
y2_lmk = np.concatenate((y2, y1[landmark_idx]))
# Make our landmarks vector, which is nan where we have no landmark information.
#
landmarks = np.stack(
[np.array([np.nan, np.nan])]*x2.shape[0] + list(
p_embedder.transform(
x1[landmark_idx]
)
)
)
# Set landmark loss weight and continue training our Parametric UMAP model.
#
p_embedder.landmark_loss_weight = 0.01
p_embedder.fit(x2_lmk, landmark_positions=landmarks)
p_emb2_x2 = p_embedder.transform(x2)
# Check how x1 looks when embedded in the space retrained on x2 and landmarks.
#
p_emb2_x1 = p_embedder.transform(x1)
绘制所有不同的嵌入结果进行比较
fig, axs = plt.subplots(3, 2, figsize=(16, 24), sharex=True, sharey=True)
axs[0,0].scatter(
emb_x1[:, 0], emb_x1[:, 1], c=y1, cmap='Spectral', s=2, alpha=0.2,
)
axs[0,0].set_ylabel('UMAP Embedding', fontsize=20)
axs[0,1].scatter(
emb_x2[:, 0], emb_x2[:, 1], c=y2, cmap='Spectral', s=2, alpha=0.2,
)
axs[0,1].scatter(
emb_x2[y2==N][:,0], emb_x2[y2==N][:,1], c='k', s=2, alpha=0.5,
)
axs[1,0].scatter(
p_emb_x1[:, 0], p_emb_x1[:, 1], c=y1, cmap='Spectral', s=2, alpha=0.2,
)
axs[1,0].set_ylabel('Initial P-UMAP Embedding', fontsize=20)
axs[1,1].scatter(
p_emb_x2[:, 0], p_emb_x2[:, 1], c=y2, cmap='Spectral', s=2, alpha=0.2,
)
axs[1,1].scatter(
p_emb_x2[y2==N][:,0], p_emb_x2[y2==N][:,1], c='k', s=2, alpha=0.5
)
axs[2,0].scatter(
p_emb2_x1[:, 0], p_emb2_x1[:, 1], c=y1, cmap='Spectral', s=2, alpha=0.2,
)
axs[2,0].set_ylabel('Updated P-UMAP Embedding', fontsize=20)
axs[2,0].set_xlabel(f'x1, No {N}s', fontsize=20)
axs[2,1].scatter(
p_emb2_x2[:, 0], p_emb2_x2[:, 1], c=y2, cmap='Spectral', s=2, alpha=0.2,
)
axs[2,1].scatter(
p_emb2_x2[y2==N][:,0], p_emb2_x2[y2==N][:,1], c='k', s=2, alpha=0.5,
)
axs[2,1].set_xlabel('x2, All Classes', fontsize=20)
plt.tight_layout()
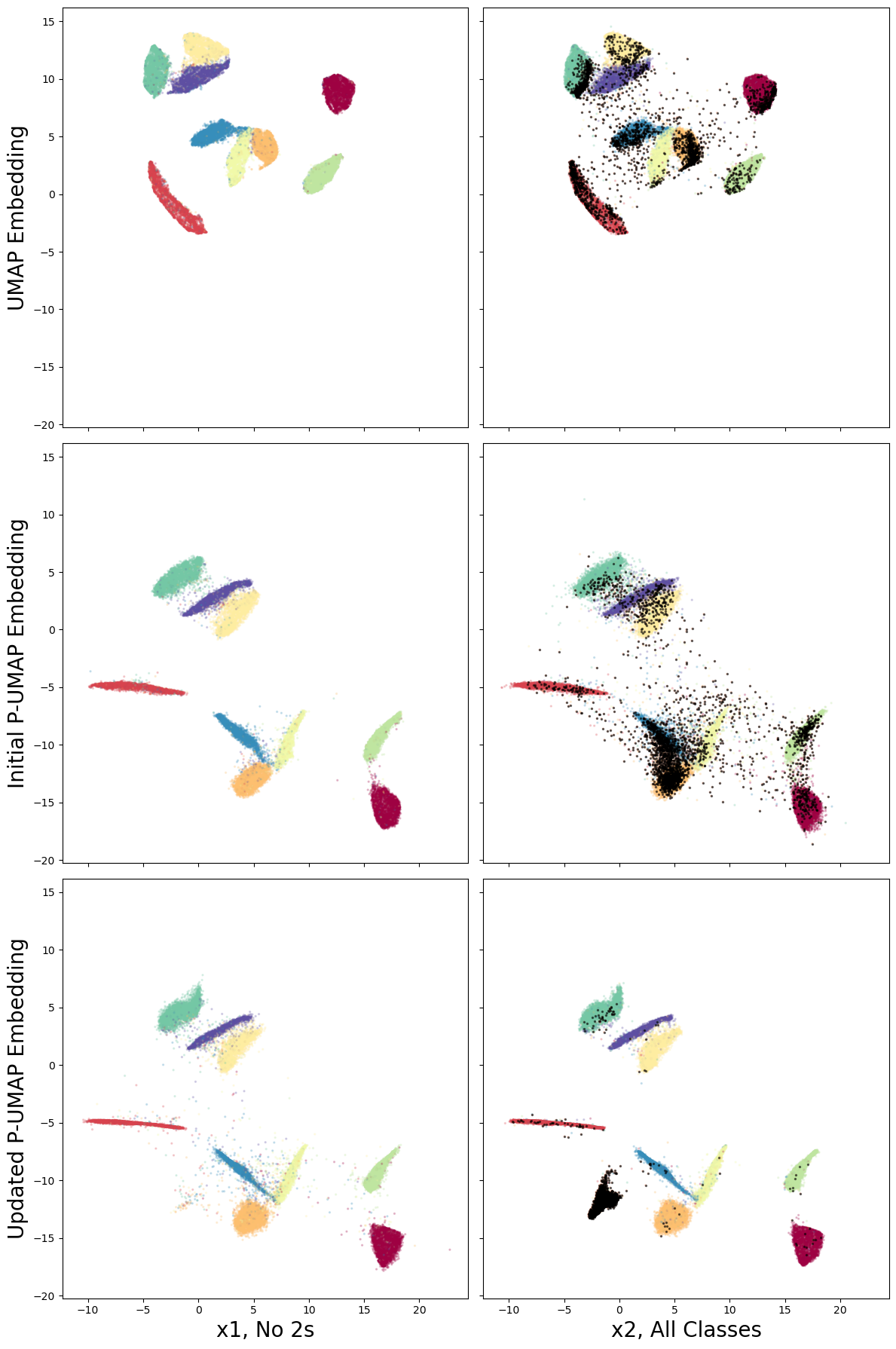
在这里我们看到我们的方法是成功的,嵌入空间保持了一致性,并且我们现在有了新类别(数字 2)的清晰聚类。这个新聚类出现在嵌入空间中合理的部分,并且其余结构得到了保留。
在此值得仔细检查 landmark loss 是否限制性太强,我们仍然希望获得良好的 UMAP 结构。为此,我们可以查看嵌入器的历史记录,它会保留重新训练步骤中的历史。
plt.plot(p_embedder._history['loss'])
plt.ylabel('Loss')
plt.xlabel('Epoch')
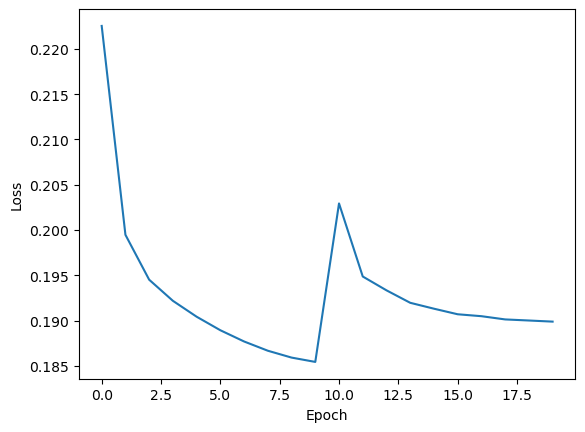
我们可以识别出在引入 x2 时损失出现的尖峰,并确认由此产生的损失与我们在 x1 上的初始训练损失相当。这告诉我们模型在 UMAP loss 和 landmark loss 之间不必做出太多妥协。如果不是这样,可以通过降低嵌入器对象的 landmark_loss_weight 属性来潜在地改进。在空间的 一致性 和最小化 UMAP loss 之间需要权衡,但关键是我们在嵌入空间中拥有平滑的变化,这将使下游任务更容易调整。在这种情况下,我们可能可以提高 landmark_loss_weight 以保持空间更一致。
除了 landmark_loss_weight 之外,还有许多其他选项可供我们尝试在此示例或其他示例上获得更好的结果。
使用原始数据(在本例中为
x1)中更大比例的点继续训练。并非所有这些点都需要作为地标,但它们可以有助于在高维空间中形成一致的图结构。改变
landmark_loss_fn。例如,如果我们想允许点在必要时移动,我们可以截断默认的欧几里得损失函数,允许隐喻的橡皮筋在某个点断开,并在发现坚持地标位置不正确时优先考虑良好的 UMAP 结构。更智能地选择地标点,例如使用 apricot-select 等包进行子模优化,或者从像 HDBSCAN 这样的分层聚类的不同部分选择点。