降维实现的性能比较
不同的降维技术可能具有截然不同的计算复杂度。除了算法本身,实现方式也会产生影响。这两个因素对实际运行给定降维算法所需的时间有着重要作用。此外,您尝试处理的数据性质也会产生影响——这主要涉及原始数据的维度。在这里,我们将简要介绍一些降维实现的性能特征。
首先,让我们加载所需的基本工具——显然包括 numpy 和 pandas,但也需要用于获取和重采样数据的工具,以及 time 模块以便进行一些基础的性能测试。
import numpy as np
import pandas as pd
from sklearn.datasets import fetch_openml
from sklearn.utils import resample
import time
接下来,我们需要实际的降维实现。为了便于解释,我们主要使用 scikit-learn,但为了比较,我们还将包括 MulticoreTSNE 对 t-SNE 的实现,其性能明显优于当前的 scikit-learn t-SNE。
from sklearn.manifold import TSNE, LocallyLinearEmbedding, Isomap, MDS, SpectralEmbedding
from sklearn.decomposition import PCA
from MulticoreTSNE import MulticoreTSNE
from umap import UMAP
接下来,我们需要绘图工具,当然还有一些数据来处理。对于这次性能比较,我们将默认使用流形学习的现行标准基准测试:MNIST 数字数据集。我们可以使用 scikit-learn 的 fetch_mldata 来为我们获取数据。
import matplotlib.pyplot as plt
import seaborn as sns
%matplotlib inline
sns.set(context='notebook',
rc={'figure.figsize':(12,10)},
palette=sns.color_palette('tab10', 10))
mnist = fetch_openml('Fashion-MNIST', version=1)
现在是时候开始考察性能了。首先,让我们看看性能如何随数据集大小的增加而变化。
性能随数据集大小的变化
随着数据集大小的增加,给定降维算法的运行时间会以不同的速率增加。如果您想在更大的数据集上运行您的算法,您不仅会关心在单个小型数据集上的比较运行时间,还会关心随着数据集变大,性能如何扩展。我们可以通过从 MNIST 数字中进行子采样(通过 scikit-learn 便捷的 resample 工具)并查看不同大小子样本的运行时间来模拟这种情况。由于此处涉及一些随机性(子样本选择和某些具有随机方面的算法),我们将希望对每种数据集大小运行几个示例。我们可以轻松地将所有这些打包在一个简单的函数中,该函数给定一个算法,将返回一个包含数据集大小和运行时间的便捷 pandas 数据框。
def data_size_scaling(algorithm, data, sizes=[100, 200, 400, 800, 1600], n_runs=5):
result = []
for size in sizes:
for run in range(n_runs):
subsample = resample(data, n_samples=size)
start_time = time.time()
algorithm.fit(subsample)
elapsed_time = time.time() - start_time
del subsample
result.append((size, elapsed_time))
return pd.DataFrame(result, columns=('dataset size', 'runtime (s)'))
现在我们只需对各种降维实现都运行这个函数,以便查看结果。由于我们不知道这些运行可能需要多长时间,我们将从一个非常小的样本集开始,只扩展到 1600 个样本。
all_algorithms = [
PCA(),
UMAP(),
MulticoreTSNE(),
LocallyLinearEmbedding(),
SpectralEmbedding(),
Isomap(),
TSNE(),
MDS(),
]
performance_data = {}
for algorithm in all_algorithms:
alg_name = str(algorithm)
if 'MulticoreTSNE' in alg_name:
alg_name = 'MulticoreTSNE'
else:
alg_name = alg_name.split('(')[0]
performance_data[alg_name] = data_size_scaling(algorithm, mnist.data, n_runs=3)
现在让我们绘制结果,以便了解情况。我们将使用 seaborn 的回归图来插值有效的缩放比例。
for alg_name, perf_data in performance_data.items():
sns.regplot('dataset size', 'runtime (s)', perf_data, order=2, label=alg_name)
plt.legend();
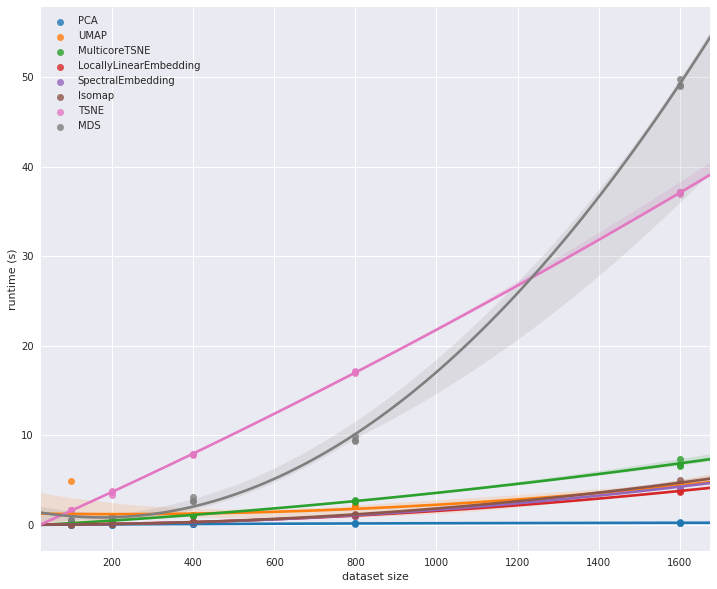
我们可以立即看到这里有一些异常值。scikit-learn 的 t-SNE 明显比大多数其他算法慢得多。然而,它不像 MDS 那样具有可伸缩性;对于更大的数据集,MDS 将很快变得完全无法处理。同时,MulticoreTSNE 表明 t-SNE 可以相当有效地运行。除了 PCA 是迄今为止最快的选项之外,很难判断其他实现的情况。要了解更多,我们需要查看在更大数据集上的运行时间。MDS 和 scikit-learn 的 t-SNE 都需要太长时间运行,所以让我们将范围限制在性能最快的实现,看看当我们扩展到更大数据集时会发生什么。
fast_algorithms = [
PCA(),
UMAP(),
MulticoreTSNE(),
LocallyLinearEmbedding(),
SpectralEmbedding(),
Isomap(),
]
fast_performance_data = {}
for algorithm in fast_algorithms:
alg_name = str(algorithm)
if 'MulticoreTSNE' in alg_name:
alg_name = 'MulticoreTSNE'
else:
alg_name = alg_name.split('(')[0]
fast_performance_data[alg_name] = data_size_scaling(algorithm, mnist.data,
sizes=[800, 1600, 3200, 6400, 12800], n_runs=3)
for alg_name, perf_data in fast_performance_data.items():
sns.regplot('dataset size', 'runtime (s)', perf_data, order=2, label=alg_name)
plt.legend();
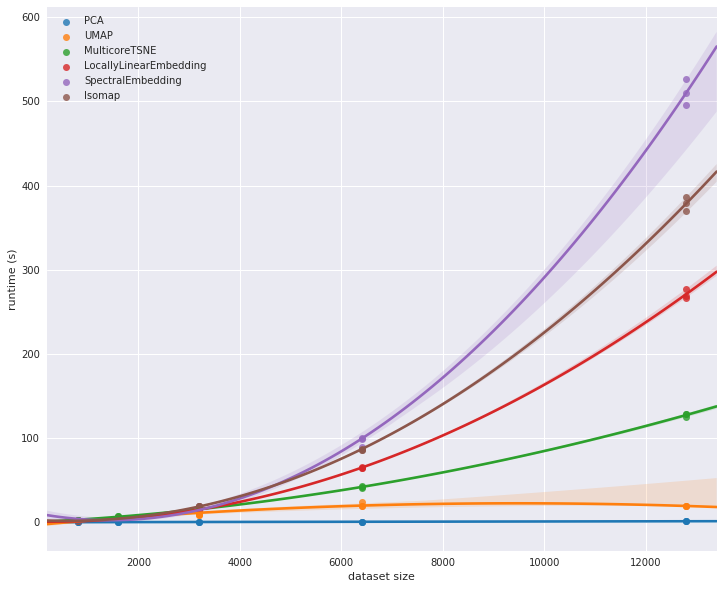
在这一点上,我们开始看到不同实现之间的一些显著差异。在之前的图中,MulticoreTSNE 看起来比其他一些算法要慢,但当我们扩展到更大的数据集时,我们看到它的相对缩放性能远优于 scikit-learn 中 Isomap、谱嵌入和局部线性嵌入的实现。
也许值得进一步扩展——直到完整的 MNIST 数字数据集。为了在合理的时间内完成,我们将不得不将注意力限制在更小的实现子集上。我们将精简到只剩下 MulticoreTSNE、PCA 和 UMAP。
very_fast_algorithms = [
PCA(),
UMAP(),
MulticoreTSNE(),
]
vfast_performance_data = {}
for algorithm in very_fast_algorithms:
alg_name = str(algorithm)
if 'MulticoreTSNE' in alg_name:
alg_name = 'MulticoreTSNE'
else:
alg_name = alg_name.split('(')[0]
vfast_performance_data[alg_name] = data_size_scaling(algorithm, mnist.data,
sizes=[3200, 6400, 12800, 25600, 51200, 70000], n_runs=2)
for alg_name, perf_data in vfast_performance_data.items():
sns.regplot('dataset size', 'runtime (s)', perf_data, order=2, label=alg_name)
plt.legend();
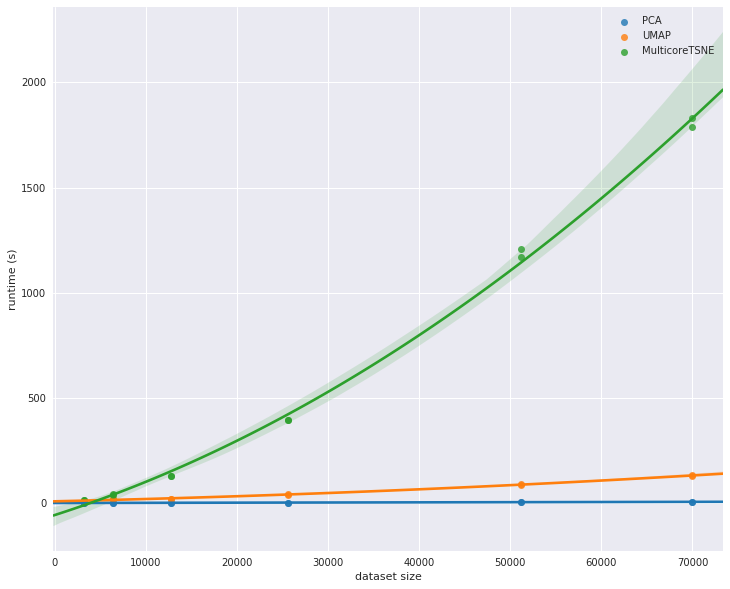
在这里,我们看到 UMAP 相对于 t-SNE 的优势真正凸显出来。虽然 UMAP 明显比 PCA 慢,但其缩放性能显著优于 MulticoreTSNE,对于更大的数据集,这种差异只会继续扩大。
关于性能随数据集大小变化的探讨到此结束。简短总结是:PCA 是迄今为止最快的选择,但为了这种速度,您可能会牺牲很多。UMAP 虽然在速度上无法与 PCA 竞争,但在此处探讨的实现中,它在性能方面显然是次优的选择。考虑到 UMAP 能够提供的结果质量,我们认为它显然是降维的一个好选择。