预计算 k-nn
本教程的目的是探讨一些使用 precomputed_knn 可能有用的情况,然后讨论如何通过它获得可重现的结果。
实际应用
尝试不同参数的 UMAP
让我们看看如何使用 precomputed_knn 来节省时间。首先,我们将在 MNIST 数据集上进行测试,该数据集包含 70,000 个样本,每个样本有 784 维。如果我们要测试一系列 n_neighbors 和 min_dist 参数,可能会花费相当多的时间重新计算数据的 knn 矩阵。取而代之的是,我们可以为我们希望分析的最大 n_neighbors 值计算 knn,然后将该 precomputed_knn 提供给 UMAP。UMAP 会自动将其修剪到正确的 n_neighbors 值,并跳过最近邻步骤,从而为我们节省大量时间。
我们注意到,为了利用 UMAP 的并行化并加速计算,我们没有使用随机状态。
from sklearn.datasets import fetch_openml
import numpy as np
import umap
import umap.plot
from umap.umap_ import nearest_neighbors
data, labels = fetch_openml('mnist_784', version=1, return_X_y=True)
labels = np.asarray(labels, dtype=np.int32)
n_neighbors = [5, 50, 100, 250]
min_dists = [0, 0.2, 0.5, 0.9]
normal_embeddings = np.zeros((4, 4, 70000, 2))
precomputed_knn_embeddings = np.zeros((4, 4, 70000, 2))
%%time
# UMAP run on the grid of parameters without precomputed_knn
for i, k in enumerate(n_neighbors):
for j, dist in enumerate(min_dists):
normal_embeddings[i, j] = umap.UMAP(n_neighbors=k,
min_dist=dist,
).fit_transform(data)
print("\033[1m"+"Time taken to compute UMAP on grid of parameters:\033[0m")
Time taken to compute UMAP on grid of parameters: Wall time: 31min 57s
%%time
# UMAP run on list of n_neighbors without precomputed_knn
# We compute the knn for max(n_neighbors)=250
mnist_knn = nearest_neighbors(data,
n_neighbors=250,
metric="euclidean",
metric_kwds=None,
angular=False,
random_state=None,
)
# Now we compute the embeddings for the grid of parameters
for i, k in enumerate(n_neighbors):
for j, dist in enumerate(min_dists):
precomputed_knn_embeddings[i, j] = umap.UMAP(n_neighbors=k,
min_dist=dist,
precomputed_knn=mnist_knn,
).fit_transform(data)
print("\033[1m"+"Time taken to compute UMAP on grid of parameters with precomputed_knn:\033[0m")
Time taken to compute UMAP on grid of parameters with precomputed_knn: Wall time: 17min 54s
使用 precomputed_knn,我们将计算时间缩短了一半!观察到我们一半的 n_neighbors 值相对较小。如果这些值的分布更高,节省的时间会更大。此外,我们还可以通过先用 n_neighbors 值为 250 的方式正常计算 UMAP,然后从该 UMAP 对象中提取 k-nn 图来节省更多时间。
通过这种方式,我们可以轻松地可视化 n_neighbors 参数如何影响我们的嵌入。
import matplotlib.pyplot as plt
fig, axs = plt.subplots(4, 4, figsize=(20, 20))
for i, ax_row in enumerate(axs):
for j, ax in enumerate(ax_row):
ax.scatter(precomputed_knn_embeddings[i, j, :, 0],
precomputed_knn_embeddings[i, j, :, 1],
c=labels / 9,
cmap='tab10',
alpha=0.1,
s=1,
)
ax.set_xticks([])
ax.set_yticks([])
if i == 0:
ax.set_title("min_dist = {}".format(min_dists[j]), size=15)
if j == 0:
ax.set_ylabel("n_neighbors = {}".format(n_neighbors[i]), size=15)
fig.suptitle("UMAP embedding of MNIST digits with grid of parameters", y=0.92, size=20)
plt.subplots_adjust(wspace=0.05, hspace=0.05)

我们看到在这种情况下,嵌入对于 n_neighbors 的选择是稳健的,而较低的 min_dist 值只是将聚类更紧密地打包在一起。
可重现性
我们强烈建议您在使用 precomputed_knn 尝试重现结果之前,查阅文档中的 UMAP 可重现性部分。
标准情况
UMAP 开箱即用,支持使用 precomputed_knn 创建可重现的结果。其工作方式与普通 UMAP 完全相同,用户可以设置一个随机种子状态,以确保结果能够精确重现。但是,必须考虑一些重要因素。
UMAP 嵌入完全依赖于:首先,计算高维数据中的图形表示;其次,学习保留该图结构体的嵌入。回想一下,我们的图形表示基于数据的 k-nn 图。如果我们有两个不同的 k-nn 图,那么我们将自然拥有两个不同的数据图形表示。因此,只有当我们使用相同的 k-nn 图时,才能确保结果可重现。在我们的例子中,这意味着所有可重现的结果都与三个值相关
计算 k-nn 时的随机种子。
计算 k-nn 时的 n_neighbors 值。
运行 UMAP 时的随机种子。
UMAP 的两次不同运行,如果这三个值相等,则保证返回相同的结果。让我们通过一个示例来看看它是如何工作的。为此,我们将创建一些数据来处理;在 60 维空间中的三个随机团块。
y = np.random.rand(1700, 60)
X = np.concatenate((y+20, y, y-20))
synthetic_labels = np.repeat([1, 2, 3], repeats=1700)
有了数据,我们可以固定上面列出的三个参数,看看两次不同的 UMAP 运行如何给出相同的结果。为避免混淆,我们将假设 UMAP 随机种子与 knn 随机种子值相同。
import umap.plot
random_seed = 10
knn = nearest_neighbors(
X,
n_neighbors=50,
metric='euclidean',
metric_kwds=None,
angular=False,
random_state=random_seed,
)
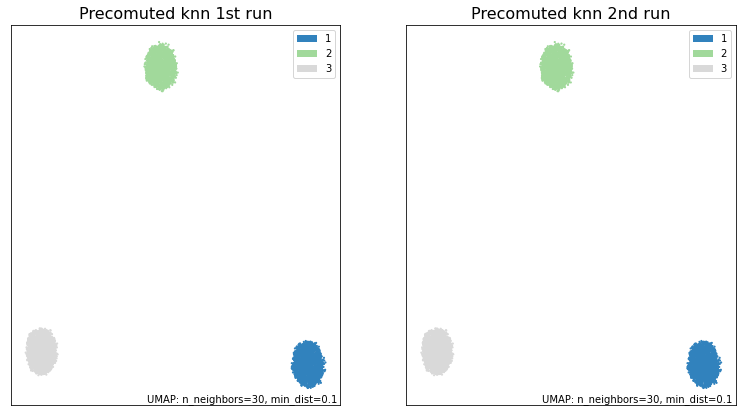
knn_umap = umap.UMAP(n_neighbors=30, precomputed_knn=knn, random_state=random_seed).fit(X)
knn_umap2 = umap.UMAP(n_neighbors=30, precomputed_knn=knn, random_state=random_seed).fit(X)
fig, ax = plt.subplots(1, 2, figsize=(13,7))
umap.plot.points(knn_umap, labels=synthetic_labels, ax=ax[0], theme='green')
umap.plot.points(knn_umap2, labels=synthetic_labels, ax=ax[1], theme='green')
ax[0].set_title("Precomuted knn 1st run", size=16)
ax[1].set_title("Precomuted knn 2nd run", size=16)
plt.show()
print("\033[1m"+"Are the embeddings for knn_umap and knn_umap2 the same?\033[0m")
print((knn_umap.embedding_ == knn_umap2.embedding_).all())
Are the embeddings for knn_umap and knn_umap2 the same? True
正如我们所见,通过固定 knn 的 random_seed 和 n_neighbors,我们能够从两次 UMAP 运行中获得完全相同的结果。相比之下,如果这些值不同,我们就不能保证结果相同。
random_seed2 = 15
# Different n_neighbors
knn3 = nearest_neighbors(
X,
n_neighbors=40,
metric='euclidean',
metric_kwds=None,
angular=False,
random_state=random_seed,
)
# Different random seed
knn4 = nearest_neighbors(
X,
n_neighbors=50,
metric='euclidean',
metric_kwds=None,
angular=False,
random_state=random_seed2,
)
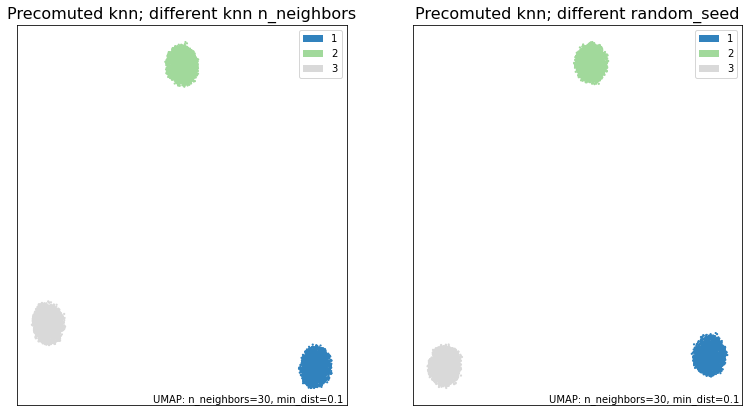
knn_umap3 = umap.UMAP(n_neighbors=30, precomputed_knn=knn3, random_state=random_seed).fit(X)
knn_umap4 = umap.UMAP(n_neighbors=30, precomputed_knn=knn4, random_state=random_seed2).fit(X)
fig, ax = plt.subplots(1, 2, figsize=(13,7))
umap.plot.points(knn_umap3, labels=synthetic_labels, ax=ax[0], theme='green')
umap.plot.points(knn_umap4, labels=synthetic_labels, ax=ax[1], theme='green')
ax[0].set_title("Precomuted knn; different knn n_neighbors", size=16)
ax[1].set_title("Precomuted knn; different random_seed", size=16)
plt.show()
print("\033[1m"+"Are the embeddings for knn_umap and knn_umap3 the same?\033[0m")
print((knn_umap.embedding_ == knn_umap3.embedding_).all())
print("\033[1m"+"Are the embeddings for knn_umap and knn_umap4 the same?\033[0m")
print((knn_umap.embedding_ == knn_umap4.embedding_).all())
Are the embeddings for knn_umap and knn_umap3 the same? False Are the embeddings for knn_umap and knn_umap4 the same? False
如果这两次运行的这三个参数不相等,我们就会得到不同的结果。
使用 precomputed_knn 重现普通 UMAP
考虑到一些额外因素,我们也可以用普通 UMAP 重现 precomputed_knn 的结果,反之亦然。和前面的情况一样,我们必须记住 k-nn 图必须相同。此外,我们还必须考虑 UMAP 如何使用我们提供的 random_seed。
如果您为 UMAP 提供 random_seed,它会将其转换为一个 np.random.RandomState (RNG)。然后使用此 RNG 固定算法中所有相关步骤的状态。需要注意的重要一点是,RNG 每次使用时都会发生变化。因此,如果我们要使用 precomputed_knn 重现结果,就必须模仿 UMAP 在调用 fit() 函数时如何操作 RNG。
有关随机状态及其行为的更多信息,请参阅 [1]。
我们将看一个如何实现此目标的示例。其他情况可以由此轻松推断。使用与之前相同的随机团块,我们尝试正常运行 UMAP,然后使用 precomputed_knn 重现结果。为此,我们必须使用 nearest_neighbors() 函数以与 fit() 相同的方式创建一个新的 k-nn 图。
from sklearn.utils import check_random_state
# First we run the normal UMAP to compare with
random_seed3 = 12
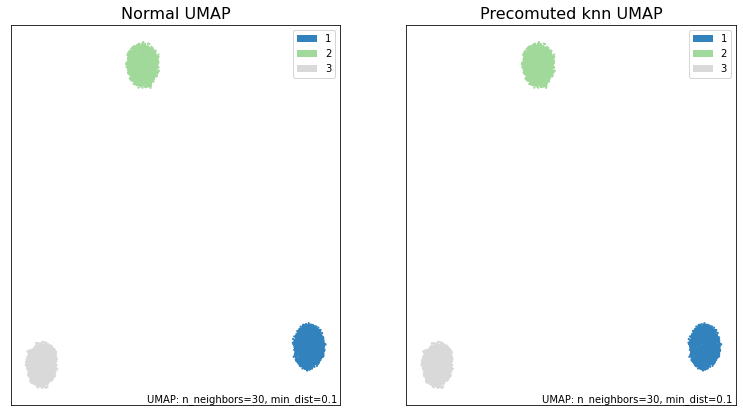
normal_umap = umap.UMAP(n_neighbors=30, random_state=random_seed3).fit(X)
# Now we run precomputed_knn UMAP
random_state3 = check_random_state(random_seed3)
# random_state3 = numpy.random.RandomState(random_seed3)
knn5 = nearest_neighbors(
X,
n_neighbors=30,
metric='euclidean',
metric_kwds=None,
angular=False,
random_state=random_state3,
)
# This mutated RNG can now be fed into precompute_knn UMAP to obtain
# the same results as in normal UMAP
knn_umap5 = umap.UMAP(n_neighbors=30,
precomputed_knn=knn5,
random_state=random_state3, # <--- This is a RNG
).fit(X)
请注意,在这种情况下,我们使用 check_random_state() 创建一个 numpy.random.mtrand.RandomState 实例,因为我们希望确保我们的 RNG 以与 UMAP 通常创建和更改它的方式完全相同的方式创建和更改。同样,我们也可以直接调用 numpy.random.RandomState()。
通过绘制和比较嵌入,我们看到我们能够获得相同的结果。
fig, ax = plt.subplots(1, 2, figsize=(13,7))
umap.plot.points(normal_umap, labels=synthetic_labels, ax=ax[0], theme='green')
umap.plot.points(knn_umap5, labels=synthetic_labels, ax=ax[1], theme='green')
ax[0].set_title("Normal UMAP", size=16)
ax[1].set_title("Precomuted knn UMAP", size=16)
plt.show()
print("\033[1m"+"Are the embeddings for normal_umap and knn_umap5 the same?\033[0m")
print((normal_umap.embedding_ == knn_umap5.embedding_).all())
Are the embeddings for normal_umap and knn_umap5 the same? True