使用 DensMAP 更好地保持局部密度
UMAP 中一个值得注意的假设是,数据均匀分布在某个流形上,而我们最终希望呈现的就是这个流形。这对于许多用例来说非常有效,但也可能存在希望保留更多关于数据相对局部密度的信息的情况。最近的一篇论文提出了一种名为 DensMAP 的技术,该技术计算局部密度的估计值,并在低维表示的优化过程中使用这些估计值作为正则化项。详细信息在该论文中有很好的解释,我们鼓励对细节感兴趣的读者阅读该论文。结果是一种低维表示,它保留了关于数据相对局部密度的信息。为了在实践中了解这意味着什么,我们加载一些模块并在一些熟悉的数据上进行尝试。
import sklearn.datasets
import umap
import umap.plot
对于测试数据,我们将使用现在很熟悉的(参见之前的教程章节)MNIST 和 Fashion-MNIST 数据集。MNIST 是一个包含 70,000 张手写数字灰度图像的集合。Fashion-MNIST 是一个包含 70,000 张时尚物品灰度图像的集合。
mnist = sklearn.datasets.fetch_openml("mnist_784")
fmnist = sklearn.datasets.fetch_openml("Fashion-MNIST")
在尝试 DensMAP 之前,我们先运行标准 UMAP,以便有一个可供比较的基准。我们将从 MNIST 数字开始。
%%time
mapper = umap.UMAP(random_state=42).fit(mnist.data)
CPU times: user 2min, sys: 15 s, total: 2min 15s
Wall time: 1min 43s
umap.plot.points(mapper, labels=mnist.target, width=500, height=500)

现在我们来尝试运行 DensMAP。在实践中,这就像在 UMAP 构造函数中添加参数 densmap=True 一样简单——这将使 UMAP 使用默认的 DensMAP 参数进行 DensMAP 正则化。
%%time
dens_mapper = umap.UMAP(densmap=True, random_state=42).fit(mnist.data)
CPU times: user 3min 42s, sys: 12.9 s, total: 3min 55s
Wall time: 2min 20s
请注意,这比标准 UMAP 稍微慢一些——需要做更多的工作。然而,值得注意的是,DensMAP 的开销相对恒定,因此当您将 DensMAP 应用于更大的数据集时,运行时间差异不会显著增加。
现在让我们看看能得到什么样的结果
umap.plot.points(dens_mapper, labels=mnist.target, width=500, height=500)
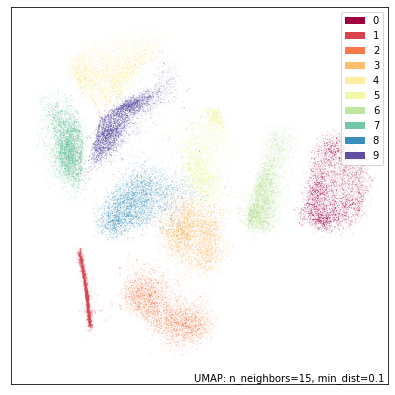
这是一个显著不同的结果——尽管数字的聚类和整体结构基本相同。最引人注目的方面是,“1”的簇被压缩成一个非常狭窄且密集的条带,而其他数字簇,尤其是“0”和“2”,则扩展开来占据了图中更多的空间。这是因为在高维空间中,“1”确实更密集地聚集在一起,主要只沿一个维度变化(“1”笔画的绘制角度)。相比之下,“0”这样的数字则有更多变化(更圆、更窄、更高、更矮、倾斜方向不同等);这导致在高维空间中局部密度较低,而 DensMAP 保留了这种局部密度的缺失。
现在我们来看看 Fashion-MNIST 数据集;像之前一样,我们先回顾一下默认 UMAP 结果是什么样子
%%time
mapper = umap.UMAP(random_state=42).fit(fmnist.data)
CPU times: user 1min 6s, sys: 8.66 s, total: 1min 15s
Wall time: 49.8 s
umap.plot.points(mapper, labels=fmnist.target, width=500, height=500)
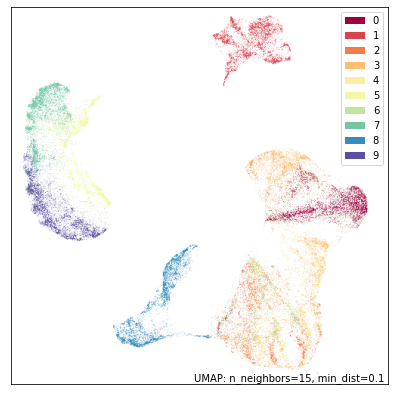
现在让我们尝试运行 DensMAP。和之前一样,这就像设置 densmap=True 标志一样简单。
%%time
dens_mapper = umap.UMAP(densmap=True, random_state=42).fit(fmnist.data)
CPU times: user 3min 48s, sys: 8.07 s, total: 3min 56s
Wall time: 2min 21s
umap.plot.points(dens_mapper, labels=fmnist.target, width=500, height=500)
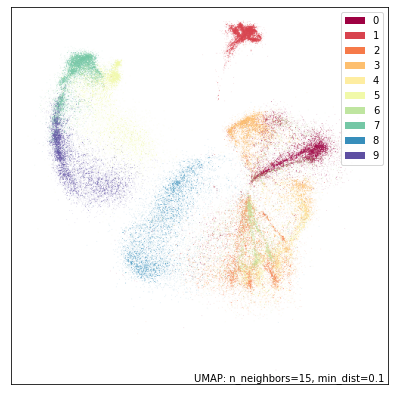
我们再次看到,DensMAP 提供了一个与 UMAP 大致相似的图,但有显著差异。在这里我们可以看到,包的簇(蓝色标记 8)实际上相当稀疏,而裤子的簇(红色标记 1)实际上非常密集,与其他类别相比变化很小。我们甚至看到了簇内部的信息。考虑靴子的簇(紫色标记 9):在其顶端相当密集,但逐渐淡入一个更加稀疏的区域。
到目前为止,我们使用了默认参数的 DensMAP,但实现提供了几个参数来精确调整如何处理局部密度正则化。我们鼓励读者查阅论文以了解许多可用参数的详细信息。对于一般用途,主要感兴趣的参数称为 dens_lambda,它控制局部密度正则化的强度。较大的 dens_lambda 值将把保持局部密度放在优先于标准 UMAP 目标的位置,而较小的值则更倾向于经典的 UMAP。默认值为 2.0。我们来稍微调整一下,以便看到其变化的效果。首先,我们将使用较高的 dens_lambda 值 5.0
%%time
dens_mapper = umap.UMAP(densmap=True, dens_lambda=5.0, random_state=42).fit(fmnist.data)
CPU times: user 3min 47s, sys: 5.04 s, total: 3min 52s
Wall time: 2min 18s
umap.plot.points(dens_mapper, labels=fmnist.target, width=500, height=500)
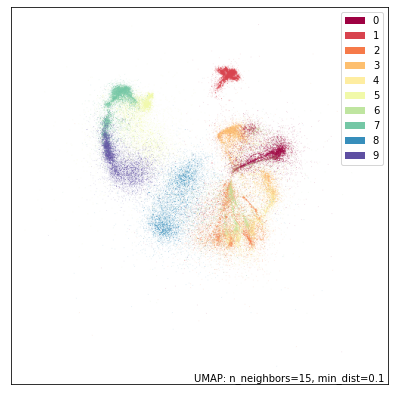
这看起来有点像我们之前看到的,但更模糊。而且……更小了?图的边界由数据设置,所以图变小反映了有一些点一直延伸到了图的边缘。这些点很可能位于高维空间中局部非常稀疏的区域,因此被推离了其他所有点。如果我们使用原始 matplotlib 和点大小更大的散点图,可以更清楚地看到这一点
fig, ax = umap.plot.plt.subplots(figsize=(7,7))
ax.scatter(*dens_mapper.embedding_.T, c=fmnist.target.astype('int8'), cmap="Spectral", s=1)
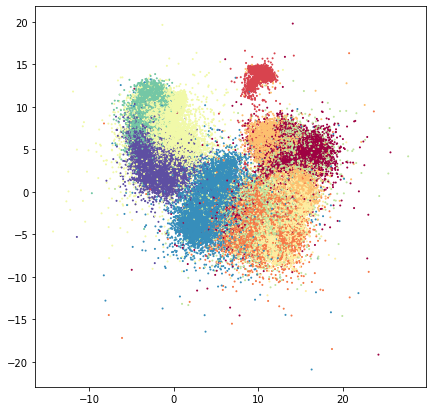
除了看到过度绘制的问题,我们实际上可以看到相当多的点在边缘周围形成了一个非常柔和的稀疏点光晕。
现在我们来尝试另一种方式,将 dens_lambda 减小到一个较小的值,这样原则上我们可以恢复一个非常接近默认 UMAP 图的结果,只是其中编码了一点局部密度信息。
%%time
dens_mapper = umap.UMAP(densmap=True, dens_lambda=0.1, random_state=42).fit(fmnist.data)
CPU times: user 3min 47s, sys: 3.78 s, total: 3min 51s
Wall time: 2min 16s
umap.plot.points(dens_mapper, labels=fmnist.target, width=500, height=500)
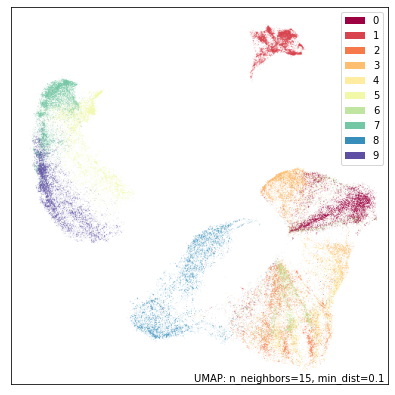
确实如此,这看起来非常像原始图,但包(蓝色标记 8)稍微更分散,而裤子(红色标记 1)则稍微更密集。这基本上是默认 UMAP,只是做了一点调整以更好地反映某种局部密度的概念。
在 Galaxy10SDSS 数据集上使用监督 DensMAP
Galaxy10SDSS 数据集是一个众包的人工标注的星系图像数据集,这些图像被分成了十个类别。DensMAP 可以学习一个部分分离数据的嵌入。为了减少运行时间,DensMAP 被应用于数据的一个子集。
import numpy as np
import h5py
import matplotlib.pyplot as plt
import umap
import os
import math
import requests
if not os.path.isfile("Galaxy10.h5"):
url = "http://astro.utoronto.ca/~bovy/Galaxy10/Galaxy10.h5"
r = requests.get(url, allow_redirects=True)
open("Galaxy10.h5", "wb").write(r.content)
# To get the images and labels from file
with h5py.File("Galaxy10.h5", "r") as F:
images = np.array(F["images"])
labels = np.array(F["ans"])
X_train = np.empty([math.floor(len(labels) / 100), 14283], dtype=np.float64)
y_train = np.empty([math.floor(len(labels) / 100)], dtype=np.float64)
X_test = X_train
y_test = y_train
# Get a subset of the data
for i in range(math.floor(len(labels) / 100)):
X_train[i, :] = np.array(np.ndarray.flatten(images[i, :, :, :]), dtype=np.float64)
y_train[i] = labels[i]
X_test[i, :] = np.array(
np.ndarray.flatten(images[i + math.floor(len(labels) / 100), :, :, :]),
dtype=np.float64,
)
y_test[i] = labels[i + math.floor(len(labels) / 100)]
# Plot distribution
classes, frequency = np.unique(y_train, return_counts=True)
fig = plt.figure(1, figsize=(4, 4))
plt.clf()
plt.bar(classes, frequency)
plt.xlabel("Class")
plt.ylabel("Frequency")
plt.title("Data Subset")
plt.savefig("galaxy10_subset.svg")
图中显示所选的数据集子集是不平衡的,但整个数据集也是不平衡的,因此本次实验仍将使用此子集。下一步是检查标准 DensMAP 算法的输出。
reducer = umap.UMAP(
densmap=True, n_components=2, random_state=42, verbose=False
)
reducer.fit(X_train)
galaxy10_densmap = reducer.transform(X_train)
fig = plt.figure(1, figsize=(4, 4))
plt.clf()
plt.scatter(
galaxy10_densmap[:, 0],
galaxy10_densmap[:, 1],
c=y_train,
cmap=plt.cm.nipy_spectral,
edgecolor="k",
label=y_train,
)
plt.colorbar(boundaries=np.arange(11) - 0.5).set_ticks(np.arange(10))
plt.savefig("galaxy10_2D_densmap.svg")
标准 DensMAP 算法无法根据星系的类型对其进行分离。监督 DensMAP 可以做得更好。
reducer = umap.UMAP(
densmap=True, n_components=2, random_state=42, verbose=False
)
reducer.fit(X_train, y_train)
galaxy10_densmap_supervised = reducer.transform(X_train)
fig = plt.figure(1, figsize=(4, 4))
plt.clf()
plt.scatter(
galaxy10_densmap_supervised[:, 0],
galaxy10_densmap_supervised[:, 1],
c=y_train,
cmap=plt.cm.nipy_spectral,
edgecolor="k",
label=y_train,
)
plt.colorbar(boundaries=np.arange(11) - 0.5).set_ticks(np.arange(10))
plt.savefig("galaxy10_2D_densmap_supervised.svg")
监督 DensMAP 确实做得更好。尽管一些类别之间有一些重叠,但原始数据集在分类上也存在一些模糊性。检查此方法的最佳方法是将测试数据投影到学习到的嵌入上。
galaxy10_densmap_supervised_prediction = reducer.transform(X_test)
fig = plt.figure(1, figsize=(4, 4))
plt.clf()
plt.scatter(
galaxy10_densmap_supervised_prediction[:, 0],
galaxy10_densmap_supervised_prediction[:, 1],
c=y_test,
cmap=plt.cm.nipy_spectral,
edgecolor="k",
label=y_test,
)
plt.colorbar(boundaries=np.arange(11) - 0.5).set_ticks(np.arange(10))
plt.savefig("galaxy10_2D_densmap_supervised_prediction.svg")
这表明学习到的嵌入可以用于新的数据集,因此此方法可能有助于检查星系图像。请在完整的 200 MB 数据集以及较新的 2.54 GB Galaxy 10 DECals 数据集上试用此方法