常见问题解答
这里汇集了一些常见问题及其解答。如果您在此处找不到您的问题,请随时在 github 上添加一个 issue。随时欢迎提出更多问题,作者将尽最大努力解答。如果您认为您有一个在此处未解答的常见问题,请在提交 issue 时建议将该问题(和解答)添加到常见问题解答中。
我应该对特征进行归一化吗?
默认答案是肯定的,但实际答案当然是“视情况而定”。如果您的特征之间存在有意义的关系(例如,经度和纬度值),那么按特征进行归一化不是一个好主意。对于本质上独立的特征,将所有特征放在(相对)相同的尺度上是有意义的。做到这一点的最佳方法是使用 scikit-learn 中的预处理工具。那里提供的所有建议都适用于 UMAP 的合理预处理,并且由于 UMAP 与 scikit-learn 兼容,您可以将所有这些整合到一个 scikit-learn pipeline 中。
我可以对 UMAP 的结果进行聚类吗?
这个问题很难回答得很好,但本质上答案是“可以,但需要谨慎”。首先,您将使用哪种聚类算法很重要。由于 UMAP 不一定会产生干净的球形簇,因此像 K-Means 这样的算法是一个糟糕的选择。我推荐使用 HDBSCAN 或类似的算法。这里的关键在于,UMAP 凭借其均匀密度假设,不能很好地保留密度。然而,UMAP 所做的是将流形的连通分量收缩到一起。只要您有足够的数据让 UMAP 区分这些信息,那么您就可以获得有用的聚类结果,因为像 HDBSCAN 这样的算法在应用 UMAP 后可以轻松地选择出这些分量。
对于聚类,UMAP 确实比 t-SNE 等算法提供了显著的改进。首先,通过保留更多的全局结构并在数据所在的流形连通分量之间创建有意义的分离,UMAP 提供了更有意义的聚类。其次,由于它支持任意嵌入维度,UMAP 允许嵌入到更大维度的空间,这使得它更适合聚类。
聚类结果都挤在一起,我看不清内部结构
UMAP 的目标之一是让点簇之间的距离具有意义。这意味着簇之间可以散开并留有相当大的空间。因此,与 t-SNE 等算法相比,簇本身在视觉上会更加紧密地堆积在一起。这是有意为之的。然而,一个问题是,许多绘图(例如 matplotlib 使用默认参数的散点图)往往只将簇显示为模糊的斑点,没有内部结构。解决这个问题实际上更多是调整绘图,而不是其他。
如果您使用 matplotlib,请考虑使用散点图中指定字形大小的参数 s。根据您的数据量,将其减小到 5 到 0.001 之间的任何值都可以产生显著的效果。bokeh 中的参数 size 也同样有用(但不需要小到这种程度)。
更一般地,特别是在大型数据集上,真正的解决方案是使用 datashader 进行绘图。Datashader 是一个绘图库,它处理散点图中的大规模数据聚合,能够更好地显示否则可能丢失的底层细节。我们强烈建议投入时间学习 datashader 进行 UMAP 绘图,特别是对于更大的数据集。
我内存不足了。求助!
对于某些数据集,近似最近邻搜索的默认选项可能导致内存过度使用。如果您的数据集不是特别大,但您发现 UMAP 在对其进行操作时内存不足,请考虑使用 low_memory=True 选项,这将切换到一种较慢但内存密集度较低的近似最近邻计算方法。这可能缓解您的问题。
UMAP 占用了我所有的核心。求助!
如果在没有随机种子的情况下运行,UMAP 将使用 numba 的并行实现进行多线程工作并使用许多核心。默认情况下,这将使用尽可能多的可用核心。如果您在共享机器上或不希望一次使用所有核心,您可以通过使用环境变量 NUMBA_NUM_THREADS 来限制 numba 使用的线程数;有关更多详细信息,请参阅 numba 文档。
是否支持 GPU 或多核 CPU?
自版本 0.4 起,提供了基本的多核支持。未来可能会添加 GPU 支持。
NVIDIA RAPIDS cuML 库中提供了 GPU 的 UMAP 实现,因此如果您需要 GPU 支持,目前这是最好的选择。
我可以添加自定义损失函数吗?
为了实现快速性能,UMAP 的 SGD 阶段已根据 UMAP 的特定需求进行了手工编码。这使得处理自定义损失函数有些困难。现在 Numba(自 0.38 版本起)支持传递函数,UMAP 的未来版本可能支持此类功能。同时,您绝对应该看看 smallvis,这是一个用于 t-SNE、LargeVis、UMAP 及相关算法的库。Smallvis 仅适用于小型数据集,但提供了更大的灵活性和控制。
是否支持 R 语言?
是的!许多人付出了努力,使 UMAP 可供 R 用户使用。
如果您想在底层使用参考实现但想要一个友好的 R 接口,那么我们推荐使用 umap,它使用 reticulate 包装了 python 代码。另一个 reticulate 接口是 umapr,但它可能不再积极开发。
如果您想要纯 R 版本,我们目前推荐使用 uwot。umap 除了其 reticulate 包装外,还提供了一个纯 R 实现。
umap 和 uwot 都可以在 CRAN 上获取。
是否有 C/C++ 实现?
据我们所知没有。目前 Numba 在提供高性能方面做得非常好,UMAP 的开发者还没有觉得需要转向更低级的语言。在某个时候可能会提供一个多线程的 C++ 实现,但目前没有时间表。
我无法让 UMAP 正常运行!
不可避免地,存在许多可能导致 UMAP 出现问题的 issue 和边缘情况。一些已知的可能导致问题的 issue 包括:
UMAP 目前不支持 32 位 Windows。这是由于该平台上的 Numba 问题造成的,不太可能很快解决。抱歉 :-(
如果您曾经使用 pip 安装了
umap包(而不是umap-learn),这可能会导致严重问题。您需要清除/删除site-packages目录中所有与 umap 相关的内容,然后重新安装umap-learn。如果当前目录中有任何名为
umap.py的文件,则会出现问题,因为会加载该文件而不是umap模块。
值得查看 github 上的 issues 页面以查找潜在的解决方案。如果所有方法都失败了,请在 github 上添加一个 issue。
PCA / UMAP / VAE 之间有什么区别?
这是流行数据集 Fashion MNIST 的一个嵌入示例。
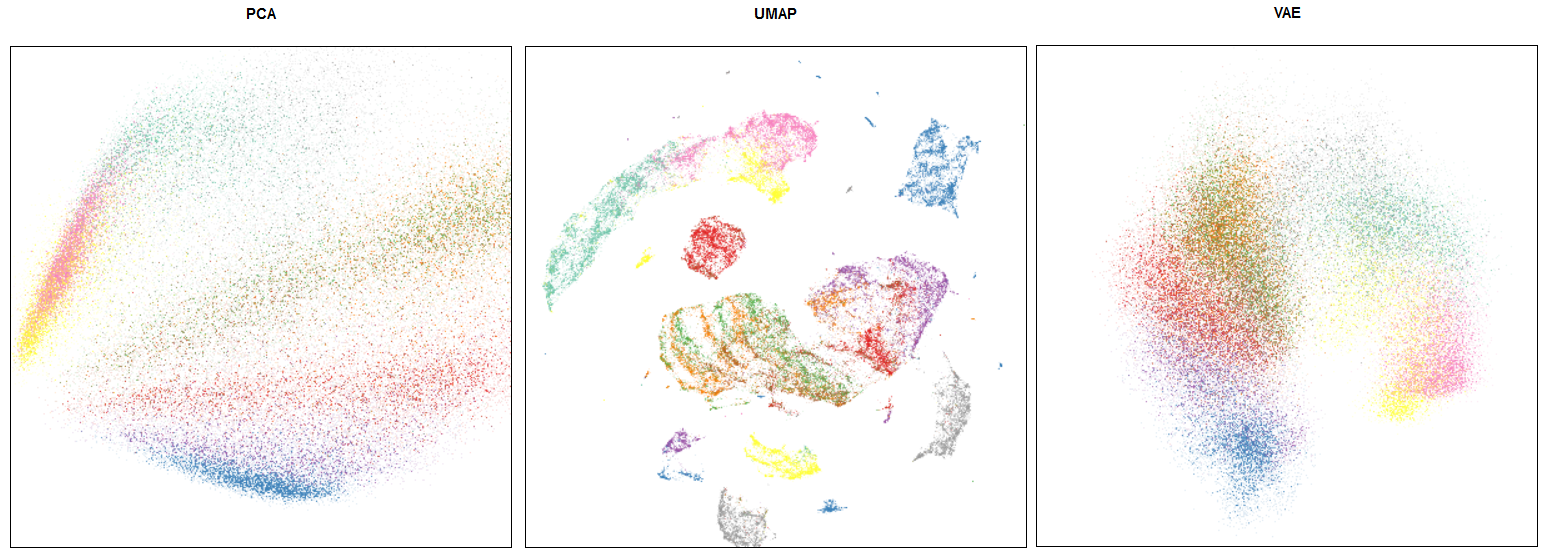
PCA / UMAP / VAE 嵌入比较
注意,FMNIST 主要是一个玩具数据集(升级版的 MNIST)。在这种简单的情况下,UMAP 显示的蒸馏结果(即如果我们在下游任务如分类中使用其嵌入)与计算成本更高的 VAEs 相当。
根据定义
PCA 是一种线性变换,您可以将其以无监督方式应用于几乎任何类型的数据。它也运行得非常快。对于大多数现实世界任务,其嵌入结果大多过于简单/无用。
VAE 是一种编码器-解码器神经网络,使用 KLD 损失和 BCE(或 MSE)损失进行训练,以强制生成的嵌入是连续的。VAE 是自编码器网络的扩展,设计上应生成不仅与实际编码数据相关,而且平滑的嵌入。
从更实用的角度来看
PCA 通常适用于现代机器上的任何合理数据集(高达数千万或数亿行);
VAEs 已被证明仅适用于玩具数据集,据我们所知,还没有适用于现实世界规模数据集(如 ImageNet)的真正有用的实际应用;
将 UMAP 应用于现实世界任务通常为下游任务(数据可视化、聚类、分类)提供了一个很好的起点,并且运行速度相当快;
考虑一个典型的 pipeline:高维嵌入 (300+) => PCA 降维到 50 维 => UMAP 降维到 10-20 维 => HDBSCAN 进行聚类 / 某些普通算法进行分类;
我应该使用哪种工具?
对于超大型或高维数据集使用 PCA(或者可以考虑寻找领域特定的矩阵分解技术,例如文本的主题建模);
对于较小数据集使用 UMAP;
VAEs 主要用于实验;
我可以在哪里了解更多?
虽然 PCA 无处不在,但您可以查看这个比较 PCA / UMAP / VAEs 的示例;
UMAP 可能出错的情况
UMAP 可能出错的一种情况是引入了与数据集中所有其他点距离最大化的数据点。换句话说,一个点的最近邻与该点的距离是最大的。一个常见的例子可能是,一个点在 Jaccard 距离下与其他任何点没有共同特征,或者一个点在连续距离函数下其最近邻的距离为 np.inf。在这两种情况下,UMAP 假定所有点都位于一个连通流形上,这可能会误导我们。从这个点的角度来看,所有其他点都是同样有效的最近邻,因此其 k 最近邻查询将返回在该最大距离处随机选择的邻居。接下来,我们将通过应用我们的 UMAP 核来归一化这个距离,该核表明一个点应该与其最近邻最大程度相似。由于所有 k 最近邻都处于相同的远距离,它们都将被我们讨论的点视为最大程度相似。当我们尝试将数据嵌入到低维空间时,我们的优化将尝试将所有这些随机选择的点拉到一起。添加足够多的此类点后,整个空间会被拉在一起,破坏我们希望识别的任何结构。
为了规避这个问题,我们在 UMAP 中添加了一个 disconnection_distance 参数,该参数将切断距离大于传入值的任何边。此参数默认为 None。当设置为 None 时,对于我们支持的任何有界度量,disconnection_distance 将设置为最大值,否则设置为 np.inf。从 UMAP 图中移除这些边将断开我们的流形,并导致这些点从它们初始化的地方开始,并通过我们的优化被推离所有其他点。
如果用户对其距离度量有很好的理解,他们可以手动设置该值,以防止其空间中特别稀疏区域的数据连接到其流形。
如果图中的顶点断开,将抛出警告消息。此时,用户可以使用 umap.utils.disconnected_vertices() 函数来识别断开的点。这可以用于过滤并重新训练一个新的 UMAP 模型,或者简单地用作可视化目的的过滤器,如下所示。
umap_model = umap.UMAP().fit(data)
disconnected_points = umap.utils.disconnected_vertices(umap_model)
umap.plot.points(umap_model, subset_points=~disconnected_points)
成功用例
UMAP 可以/已经成功应用于以下领域:
生物学中的单细胞数据可视化;
基于行为数据映射恶意软件;
预处理词组向量用于聚类;
预处理图像嵌入(Inception)用于聚类;
还有更多——如果您有成功的用例,请提交 pull request 将其添加到此列表!