使用 UMAP 转换新数据
UMAP 对于生成可视化很有用,但如果想更广泛地利用 UMAP 完成机器学习任务,重要的是能够训练一个模型,然后在后续阶段将新数据传递给模型,并让其将数据转换到学习到的空间中。例如,如果我们使用 UMAP 学习一个潜在空间,然后在转换到潜在空间的数据上训练一个分类器,那么只有当我们可以将被预测的数据转换到分类器使用的潜在空间时,该分类器才对预测有用。幸运的是,UMAP 使得这一点成为可能,尽管它比其他一些允许这样做的转换器要慢一些。
本教程将逐步介绍一个简单的案例,其中我们期望高维向量的整体分布在训练数据和测试数据之间保持一致。有关这可能出错的原因以及如何使用参数化 UMAP 修复它的更多详细信息,请参阅 使用参数化 UMAP 转换新数据。
为了演示此功能,我们将利用 scikit-learn 及其包含的数字数据集(有关数字数据集的示例,请参阅 如何使用 UMAP)。首先,让我们加载所有完成此操作所需的模块。
import numpy as np
from sklearn.datasets import load_digits
from sklearn.model_selection import train_test_split, cross_val_score
from sklearn.neighbors import KNeighborsClassifier
from sklearn.svm import SVC
import matplotlib.pyplot as plt
import seaborn as sns
%matplotlib inline
sns.set(context='notebook', style='white', rc={'figure.figsize':(14,10)})
digits = load_digits()
为了保持公正性,让我们使用 sklearn 的 train_test_split 方法来分离训练集和测试集(对不同数字类型进行分层抽样)。默认情况下,train_test_split 会将 25% 的数据划分为测试集,这在本例中似乎是合适的。
X_train, X_test, y_train, y_test = train_test_split(digits.data,
digits.target,
stratify=digits.target,
random_state=42)
现在,为了对我们正在观察的情况有一个基准概念,让我们训练几个不同的分类器,然后看看它们在测试集上的得分如何。在本例中,让我们尝试一个支持向量分类器和一个 KNN 分类器。理想情况下,我们应该调整超参数(例如使用 k 折交叉验证进行网格搜索),但为了本简单演示的目的,我们只对两个分类器使用默认参数。
svc = SVC().fit(X_train, y_train)
knn = KNeighborsClassifier().fit(X_train, y_train)
下一个重要的问题是这些分类器在测试集上的性能如何。方便的是,sklearn 提供了一个 score 方法,可以输出在测试集上的准确率。
svc.score(X_test, y_test), knn.score(X_test, y_test)
(0.62, 0.9844444444444445)
结果是支持向量分类器在本例中显然具有较差的超参数(我预计通过一些调优,我们可以构建一个更准确的模型),而 KNN 分类器表现非常好。
现在的目标是利用 UMAP 作为预处理步骤,可以将其放入管道中。因此,显然我们需要加载 umap 模块。
import umap
要将 UMAP 用作数据转换器,我们首先需要使用训练数据拟合模型。这与 如何使用 UMAP 示例中使用 fit 方法的方式完全一样。在本例中,我们只需将训练数据提供给它,它就会学习一个合适的(默认为二维)嵌入。
trans = umap.UMAP(n_neighbors=5, random_state=42).fit(X_train)
由于我们嵌入到了二维空间,我们可以将结果可视化,以确保这种方法能带来潜在的好处。这只需生成一个散点图,数据点按其所属类别着色即可。请注意,一旦我们将模型拟合到某些数据上,嵌入的训练数据就可以通过 UMAP 模型的 .embedding_ 属性进行访问。
plt.scatter(trans.embedding_[:, 0], trans.embedding_[:, 1], s= 5, c=y_train, cmap='Spectral')
plt.title('Embedding of the training set by UMAP', fontsize=24);
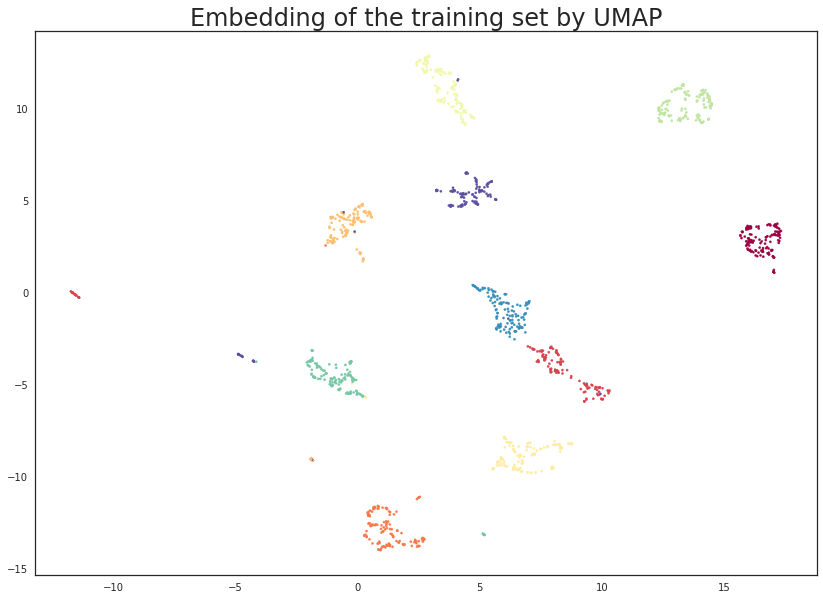
这看起来非常有希望!大多数类别都得到了非常清晰的分离,这给了我们一些希望,表明它可以帮助提高分类器的性能。值得注意的是,这是一个完全无监督的数据转换;我们可以使用训练标签信息,但这将在 后续教程 中介绍。
现在我们可以在嵌入的训练数据上训练一些新模型(同样是 SVC 和 KNN 分类器)。这看起来和之前完全一样,只是现在我们将嵌入的数据传递给它。请注意,对与模型训练时使用的输入完全相同的数据调用 transform 将简单地返回 embedding_ 属性,因此 sklearn 管道将按预期工作。
svc = SVC().fit(trans.embedding_, y_train)
knn = KNeighborsClassifier().fit(trans.embedding_, y_train)
现在我们要处理测试数据,这是任何模型(UMAP 或分类器)都没有见过的。为此,我们使用标准的 sklearn API,并利用 transform 方法,这次我们将新的、未见过的测试数据传递给它。我们将此结果赋值给 test_embedding,以便我们可以仔细查看将现有 UMAP 模型应用于新数据的结果。
%time test_embedding = trans.transform(X_test)
CPU times: user 867 ms, sys: 70.7 ms, total: 938 ms
Wall time: 335 ms
请注意,转换操作非常高效——耗时不到半秒。与其他一些转换器相比,这稍微慢一些,但对于许多用途来说已经足够快了。请注意,随着训练集和/或测试集大小的增加,性能会按比例下降。还值得注意的是,由于 Numba JIT 开销,第一次调用 transform 可能会很慢——后续运行将非常快。
下一个重要的问题是 transform 对我们的测试数据做了什么。原则上,我们得到了测试集的一个新的二维表示,理想情况下,这应该基于训练集的现有嵌入。我们可以通过可视化数据(因为我们在二维空间中)来检查这是否属实。一个简单的散点图就足够了,就像之前一样。
plt.scatter(test_embedding[:, 0], test_embedding[:, 1], s= 5, c=y_test, cmap='Spectral')
plt.title('Embedding of the test set by UMAP', fontsize=24);

结果看起来符合我们的预期;根据上面可视化的训练数据的嵌入情况,测试数据已经被嵌入到二维空间中,位置正是我们应该预期的(按类别)。这意味着我们现在可以通过将新转换的测试集交给在嵌入的训练数据上训练的模型来尝试它们。
svc.score(trans.transform(X_test), y_test), knn.score(trans.transform(X_test), y_test)
(0.9844444444444445, 0.9844444444444445)
结果相当不错。虽然 KNN 分类器的准确率没有提高,但考虑到数据,也没有太多提升空间。另一方面,SVC 的准确率已提高到与 KNN 分类器相当的水平。当然,我们可能可以通过更好地设置 SVC 超参数来达到这个准确率水平,但这里的重点是我们可以将 UMAP 用作标准的 sklearn 转换器,作为 sklearn 机器学习管道的一部分。
只是为了好玩,我们可以运行相同的实验,但这次降维到十维(我们无法再可视化)。实际上,在这种情况下这不会带来太多好处——对于数字数据集,二维对 UMAP 来说已经足够了,更多的维度不会有帮助。另一方面,对于更复杂的数据集,更多的维度可能会带来更忠实的嵌入,值得注意的是我们并不局限于仅有两个维度。
trans = umap.UMAP(n_neighbors=5, n_components=10, random_state=42).fit(X_train)
svc = SVC().fit(trans.embedding_, y_train)
knn = KNeighborsClassifier().fit(trans.embedding_, y_train)
svc.score(trans.transform(X_test), y_test), knn.score(trans.transform(X_test), y_test)
(0.9822222222222222, 0.9822222222222222)
我们看到,在这种情况下,我们的准确率分数实际上略有下降(请注意,这可能在评分的潜在噪音范围内)。然而,对于更有趣的数据集,更高维度的嵌入可能会带来显著的提升——在包含 UMAP 的管道中,将其作为网格搜索的参数之一进行探索肯定是有价值的。